La population âgée est sujette aux infections microbiennes, qui peuvent entraîner la mort. Par conséquent, il est important de comprendre pourquoi ce groupe est vulnérable aux infections microbiennes, en particulier aux infections bactériennes. une récente rapports scientifiques L’étude a lié les données de deux sources pour comprendre les déterminants de l’infection microbienne dans la population âgée du Royaume-Uni.
 Étude : Infection microbienne diagnostiquée en laboratoire chez les participants à la biobanque anglaise du Royaume-Uni par rapport à la population générale. Crédit d’image : Kateryna Kon/Shutterstock
Étude : Infection microbienne diagnostiquée en laboratoire chez les participants à la biobanque anglaise du Royaume-Uni par rapport à la population générale. Crédit d’image : Kateryna Kon/Shutterstock
Bas
La prévalence des infections bactériennes augmente significativement avec l’âge. Selon les données de surveillance britanniques, l’incidence des Escherichia coli (E. coli) est environ dix fois plus élevée chez les hommes âgés de 45 à 64 ans et environ 100 fois plus élevée chez les hommes âgés de 75 ans et plus par rapport au groupe d’âge le plus jeune, c’est-à-dire les 15-44 ans. Des tendances similaires ont été observées avec Staphylococcus aureus, pneumonie stéotococcique, Oui Streptococcus pyogènes bactéries
Actuellement, il n’y a pas d’explication claire pour laquelle les personnes âgées sont plus vulnérables aux infections microbiennes. Cependant, les facteurs de risque environnementaux, tels que la nutrition, le mode de vie et le logement, ont été considérés comme des facteurs contributifs possibles. De plus, les niveaux de protéine C-réactive (CRP) pourraient contribuer au risque d’infection individuel.
Des études sérologiques ont indiqué que le vieillissement est associé à une diminution progressive de l’immunité adaptative, c’est-à-dire des réponses des lymphocytes T et des taux d’anticorps, entraînant une augmentation des pneumonies à pneumocoque et des infections à zona. .
En plus de l’imagerie radiologique, l’échantillonnage microbiologique (p. ex., sang, urine, crachats, liquide péritonéal et liquide céphalo-rachidien) peut également être utilisé pour diagnostiquer une infection en identifiant l’organisme responsable de l’infection. En Angleterre, les échantillons microbiologiques sont normalement traités dans les laboratoires hospitaliers relevant du National Health Service.
À propos de l’étude
La présente étude a utilisé une cohorte de population à grande échelle, à savoir la UK Biobank (UKB), pour comprendre les déterminants de l’infection bactérienne et son influence sur les problèmes de santé ultérieurs.
UKB est une cohorte prospective contenant des informations sur environ 500 000 hommes et femmes âgés de 40 à 69 ans entre 2006 et 2010. Initialement, cette cohorte a été conçue pour évaluer les déterminants environnementaux et génétiques conduisant à des maladies mortelles courantes.
Public Health England (PHE) a mis en place un système de surveillance de deuxième génération (SGSS) pour surveiller et améliorer la santé publique. L’ensemble de données SGSS contient des informations régulièrement mises à jour sur les agents pathogènes humains, tels que Campylobacter, Salmonelleet d’autres agents pathogènes d’origine alimentaire. En outre, il contient des rapports de tests antimicrobiens contre des agents pathogènes importants.
L’étude actuelle a démontré la possibilité de relier les données de cohorte prospective UKB avec un ensemble de données national contenant des informations sur les cultures microbiennes en Angleterre (SGSS).
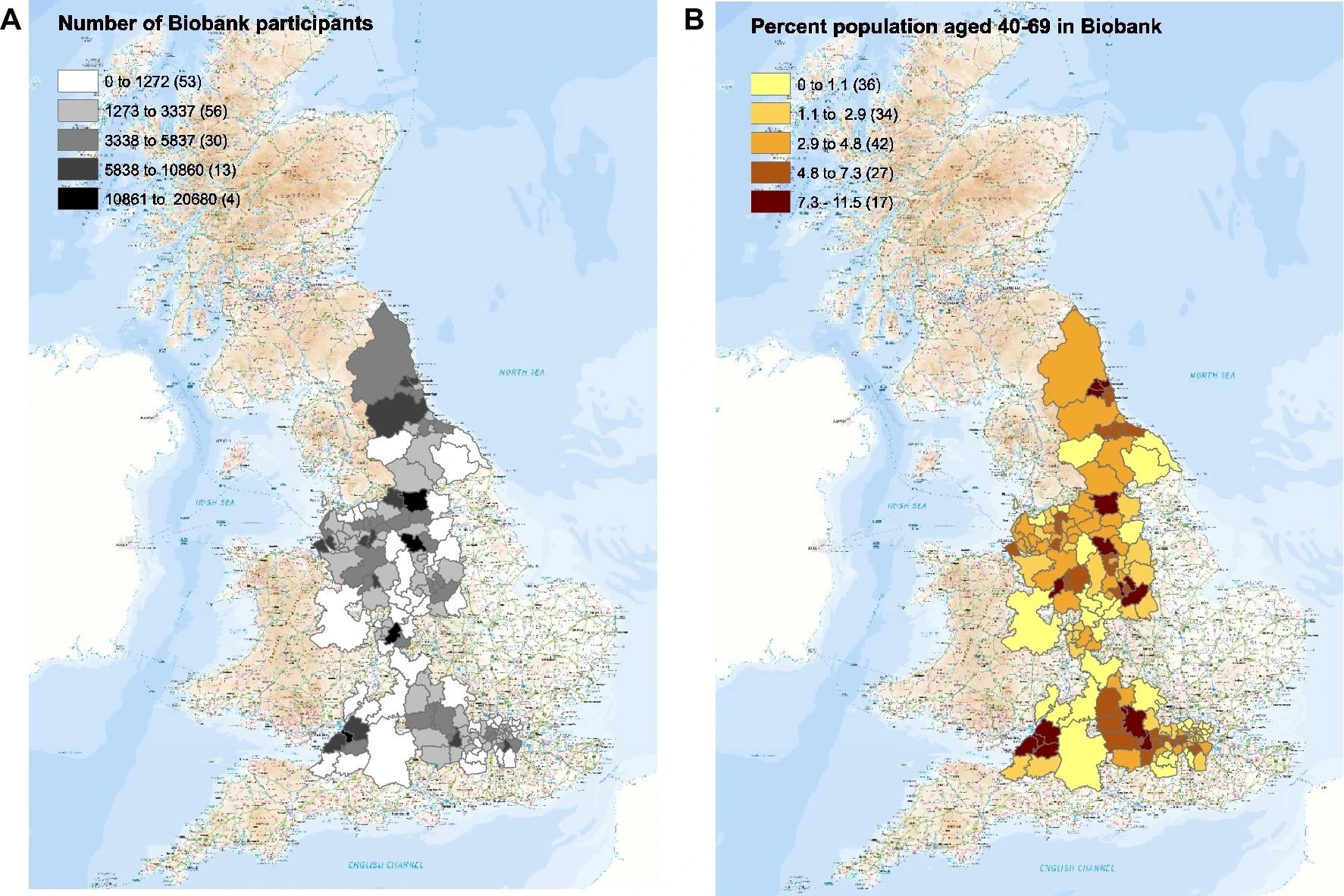 Le nombre de participants à la UK Biobank identifiés dans chaque autorité locale où le recrutement a eu lieu (à gauche) et le pourcentage de la population recrutée dans chaque autorité locale (à droite). Les cartes ont été réalisées avec ARCGIS par UKHSA.
Le nombre de participants à la UK Biobank identifiés dans chaque autorité locale où le recrutement a eu lieu (à gauche) et le pourcentage de la population recrutée dans chaque autorité locale (à droite). Les cartes ont été réalisées avec ARCGIS par UKHSA.
Résultats de l’étude
Un total de 4 726 417 échantillons provenant de 4 066 974 personnes ont été identifiés entre le 1er avril 2010 et le 30 juin 2016. En particulier, l’ensemble de données SGSS a une couverture presque complète de l’Angleterre à partir de 2015, à partir de sources de soins primaires et secondaires. Cependant, les données microbiennes antérieures à 2015 n’étaient disponibles que dans certaines régions d’Angleterre.
E. coli Oui S. aureus étaient les agents pathogènes les plus courants dans la cohorte de l’étude. Cependant, les agents pathogènes couramment trouvés dans la plupart des communautés et des hôpitaux étaient Streptocoque spp., entérocoque sp, pseudomonas aerugineux, Oui entérobactéries. Le groupe d’âge plus âgé s’est avéré plus sensible aux infections des voies urinaires, ainsi qu’aux infections respiratoires, cutanées / plaies et intestinales. La prévalence de E. coli était plus élevé chez les femmes que chez les hommes, tandis que S. aureus a montré le schéma inverse.
Points forts de la liaison d’enregistrements
Le suivi des isolats microbiologiques de plus de 450 000 individus présente de nombreuses opportunités de recherche. Premièrement, étant donné que UKB contient des données génomiques sur l’hôte, le facteur génétique qui augmente le risque d’infection microbienne dans le groupe d’âge plus avancé peut être étudié.
La mise en relation des enregistrements UKB et SGSS sera l’occasion de mieux appréhender le concept de protection de la nature. Cette plate-forme offre une possibilité importante de surveiller la résistance aux antimicrobiens. Les progrès technologiques permettront d’identifier les facteurs génétiques microbiens associés à la virulence et à la résistance aux antibiotiques.
La possibilité d’intégrer les données microbiologiques de l’ensemble de données SGSS avec UKB, c’est-à-dire le couplage d’enregistrements, est d’une grande importance dans la santé publique et la recherche biomédicale.
Limites actuelles de liaison d’enregistrements
L’approche actuelle consistant à relier les données UKB et SGSS présente plusieurs limites. Par exemple, UKB ne représente pas entièrement la population anglaise. De plus, ceux qui se sont inscrits à des études basées sur le volontariat étaient en meilleure santé et plus riches que la population générale du Royaume-Uni. En raison de «l’effet volontaire sain», les taux d’isolement microbien étaient significativement faibles chez les participants à l’UKB par rapport aux personnes du même âge dans la population générale du Royaume-Uni.
Une différence systématique dans l’utilisation et la sélection d’antibiotiques a également été observée entre UKB et d’autres cohortes de population. Malgré le manque de représentativité, ces biais peuvent être quantifiés et les données peuvent être examinées efficacement pour les associations exposition-maladie.
Malgré les méthodes standardisées suivies par SGSS, l’hétérogénéité entre les protocoles microbiens et les technologies dans les laboratoires peut présenter une certaine variation dans les résultats. De plus, la détermination d’une infection microbiologique dépend de l’accès à un centre médical où des échantillons viables sont prélevés. Le prélèvement microbien dépend aussi du médecin.
- Hilton, B. et al.. (2023) Infection microbienne diagnostiquée en laboratoire chez les participants à la biobanque anglaise du Royaume-Uni par rapport à la population générale. rapports scientifiques, 13 496.


